Introduction
인삼 (Panax ginseng)은 농가 소득 증대를 위한 주요 작물 중 한 품목으로서 국내 재배 면적이 22,165 ha (KOSIS, 2019)로 중북부지방에 걸쳐 경작되고 있다. 특히 생산된 인삼 상당부분은 수출되고 있어 농가소득원으로서의 가치가 높은 작물이다. 인삼은 다년생 및 숙근성 작물로 평균 재배 기간은 약 3 - 5년으로 타작물에 비해 상대적으로 긴 편이며, 저온 다습한 동일 포장 내 장기간 연작으로 인해 토양전염성병원균 (Soil-borne disease pathogen)의 노출에 아주 취약한 편이다 (Rahman and Punja, 2007). 인삼 경작 시 발생하는 병해의 상당 부분은 식물병원성 곰팡이와 같은 토양유래병원균에 의해서 발병되는 것으로 알려져 있다. 그 중 인삼잘록병은 대표적인 병원균으로서 Pythium solani, Pythium ultimum, Rhizoctonia solani에 의해 주로 발병되며, 대부분 고년생 (4 - 6년생) 인삼에서 관찰되는 질병으로 전국 인삼재배 지역 (최대 발병주율 18% 수준)에 심각한 피해를 입히고 있다.
인삼잘록병 방제를 위한 관행적 방안으로 경종적 측면에서는 답전 윤환의 논삼재배법이 권고되고 있고, 직접 토양을 훈증처리 (Lee et al., 2018) 하거나, Fludioxonil 계열의 살균제처리에 의한 화학적 방제 (Choi et al., 2009)가 실시되고 있다. 하지만 관행방제법을 활용할 경우 뿌리썩음병 원인균으로 밝혀진 식물병원성 곰팡이들은 불리한 토양 환경 내에서는 후막포자를 형성하여 장기간 생존이 가능하기 때문에 근본적 방제의 어려움이 있다. 토양 훈증제의 사용은 토양 내 유용미생물들까지 사멸시킬 수 있기 때문에 토양 생태계의 파괴를 초래할 수 있을 뿐만 아니라 식품안정성 및 다양한 환경오염 문제를 유발 시킬 수 있다 (Shin et al., 2012). 최근 합성 농약 사용 규제 정책 및 친환경 농자재를 이용한 농산물 생산에 대한 관심이 꾸준하게 증가하고 있어 친환경적 생물방제 기술에 대한 수요가 늘어나고 있는 추세에 있다. 하지만 급증하는 관심사에 비해 친환경방제기술에 대한 연구는 여전히 초기 연구 수준에 머무르고 있어 관련 기술 개발이 시급한 실정이다.
유용미생물을 활용한 식물병의 생물학적 주요 방제 기작은 ① 유용균의 길항 물질 생성에 의한 토양 내 병원균의 활성 억제, ② 식물유도저항성 (ISR) 생성에 의한 식물병활성 억제, ③ 토양서식지 내 양분 경쟁에 의한 병원균 억제 전략으로 요약될 수 있다 (Lugtenberg and Kamilova, 2009). 특히 토양 등에서 분리되는 Bacillus subtilis group을 비롯한 Bacillus velezensis group은 스트레스가 많은 환경 조건에서 내생포자 (Endospore) 형성하여 장기간 휴면할 수 있어, 극한환경에서의 적응력 및 생존력이 뛰어난 편이다. 특히 타 미생물과 다르게 항생물질 생산능력이 뛰어나 병원성 곰팡이의 활성을 억제할 수 있는 주요 방제 미생물로서 그 활용 가치가 매우 높은 편이다.
실제로 Bacillus velezensis GH1-13을 포함한 Bacillus subtilis group의 생육촉진, 항균 활성 및 길항 대사물질 관련 연구가 국내외에서 활발히 진행되고 있으며, 이를 활용한 식물병 방제 사례 및 기작이 보고 되고 있다 (Chung and Kim, 1978; Chung et al., 1982; Shim and Lee, 1991; Chung et al., 2006; Kim et al., 2012a; Chowdhury et al., 2015; Kim et al., 2016; Kim et al., 2017a; Kim et al., 2017b). 특히 인삼재배 토양에서 분리한 Bacillus amyloliquefaciens 균주의 경우 인삼 뿌리썩음병원균 (Cylindrocarpon destructans, Fusarium oxysporum, Fusarium solani, Rhizoctonia solani, Sclerotinia nivalis)에 대해 항균 활성이 존재하는 것으로 확인되어 차후 생물학적 방제를 위한 미생물 제재로서 활용가능성이 높은 것으로 조사된 바 있으며, 관련 연구가 점차 확대되어 가고 있다 (Kim et al., 2016). Bacillus 처리에 따른 식물병 방제 효과는 주로 실내 실험 수준에서 수행되고는 있으나, 인삼 토양에 직접 균주를 처리 후 유용균 및 병원균의 밀도 변화를 조사한 연구는 많지 않다. 이로 인해 균주처리에 따른 유용균의 토양 내 정착능 및 생물학적 방제 효과가 제대로 평가되지 못하고 있다.
본 연구의 목적은 인삼잘록병 방제를 위해 기존 연구에서 선발된 균주를 토양 처리 후 1) 유용균과 병원균의 밀도 변화를 qPCR을 통하여 모니터링하고, 2) 유용미생물 처리에 따른 토양 미생물 군집 특성 변동 및 3) 토양 화학적 특성 변화를 종합적으로 평가하여, 친환경 인삼잘록병 방제 기술을 개발하고자 하였다.
Materials and Methods
마이크로코즘 실험 충청북도 음성군 소이면 인삼특작부에 위치한 인삼 재배 포장에서 토양을 채취하여, 마이크로코즘 실험을 수행하였다. 시험토양의 이화학적 특성은 pH (1:5) 6.7, 유기물함량 12 g kg-1, 유효인산 179 mg kg-1, 치환성 양이온 칼륨, 칼슘, 마그네슘 함량은 각각 0.1, 3.6, 1.1 cmol(+) kg-1 이었으며, 토성은 양질 사토 (Loamy sand)로 모래 비율이 80% 이상인 배수가 잘되는 일반적인 인삼 재배지 토양이었다. 채취한 토양은 그늘에서 풍건하여 10 mm 체 통과 후 불순물 등을 제거하였다. 동일하게 스티로폼 어상자에 충진하여 처리구 당 3반복으로 마이크로코즘을 제작한 다음 비닐하우스 내에 완전임의배치법으로 실험을 수행하였다.
균주 처리 및 토양 시료 채취 본 연구에서는 사용된 균주는 인삼 뿌리썩음병원균에 길항능력이 있는 것으로 이미 보고된 Bacillus velezensis CC112 균주를 사용하여 제재화된 시제품 (안잘록)을 사용하였다 (Lee et al., 2016). 균주 처리방법은 일반적으로 사용되고 있는 관주 처리와 종자 침지 두 가지 방법을 선택하였다. 관주 처리의 경우 CC112 균주를 제재화 한 시제품을 1% (100배 희석, 107 cfu mL-1) 수준으로 희석하여 어상자당 2 리터씩 관주하였으며, 무처리의 경우 균주 처리만 제외하고 동일하게 처리하였다. 침지의 경우 관주 처리와 동일 희석액에 1시간 동안 종자를 침지한 다음 약 2시간 정도 클린벤치에서 건조 후 파종하였고, 무처리의 경우 유용 균주 배양 때 사용하였던 희석 배양액 만을 처리하였다. 토양 시료채취는 균주 처리 후 약 3 - 4일 간격으로 실시하였으며, 균주 밀도가 점차 감소하여 무처리와 차이가 존재하지 않는 시점까지 수행하였다.
토양 DNA는 제조사의 방법을 참고하여 토양 0.5 g을 PowerSoil DNA isolation kit (Quiagen, USA)를 사용하여 추출하였다. 추출된 토양 DNA는 NanoDropTM 1000 Spectrophotometer (Thermo Fisher Scientific, USA)을 통해 정량하였다.
선발 균주 및 병원균 밀도 토양 내 처리균주 및 인삼잘록병원균의 밀도를 조사하기 위해 SYBR green을 이용한 quantitative real time PCR (qPCR) 분석을 수행하였다. 선발균주를 효과적으로 검출하기 위해 유전체 분석 데이터를 활용하여, 특이 삽입유전자부위(Insertion sequence region)를 기반으로 특이 프라이머를 제작하였다. 본 연구에서 사용된 특이 프라이머 및 병원균 검출용 프라이머 염기서열은 Table 1에 나타내었다. qPCR 반응은 GoTaq qPCR Master Mix 2X (Promega, USA) 10 uL, BSA (bovine serum albumin, 10 mg mL-1) 2 uL, 프라이머 각각 0.5 uL를 포함하여 최종 부피가 20 uL가 되도록 멸균한 증류수를 첨가하였다. qPCR은 CFX96 (Bio-Rad, USA)을 이용하여 수행되었으며, 반응조건은 특이 프라이머의 경우 95°C에서 5분 가열한 후, 95°C 15초, 63°C 30초, 72°C 30초 순으로 40회 반복하면서 Ct 값을 측정하였다. qPCR 정량 분석을 위해 표준물질을 제작하였다. CC112 및 인삼잘록병원균 특이 프라이머로 PCR을 수행한 후 최종 유전자 증폭산물을 QIAquick Gel Extraction Kit (Qiagen, USA)를 이용해 얻은 후 정제하여 E.coli에 형질전환 실시하였다. 형질전환 E.coli는 LB broth 배지에 접종 후 24시간 동안 37°C에서 배양 한 후 Wizard® Plus SV Minipreps DNA Purification Systems (Promega, USA)를 사용하여 plasmid DNA를 추출하였다. 순차적으로 plasmid DNA를 희석하여 표준곡선을 제작하였다. 표준물질을 사용하여 qPCR 반응 최적화를 실시하였고, 반응 곡선, qPCR 증폭 효율, 융해곡선을 분석하여 최적 분석 조건임을 확인하였다.
Table 1.
Primers for quantitative real-time PCR (qPCR) used in this study.
인삼 잘록병원균의 정량분석은 기존에 보고된 Nested qPCR 방법을 응용하여 수행하였다 (White et al., 1990; Hayden et al., 2004; Lievens et al., 2006). 곰팡이 전체 군집을 증폭하기 위해 ITS1와 ITS4 프라이머를 사용하여 1차적으로 곰팡이 군집 전체를 증폭하였다. 동일한 증폭 산물을 100배 희석하여 DNA 주형으로 사용하였으며, ST-RS1와 ITS4 (R. solani)와 AFP276와 ITS4 (P. ultimum) 프라이머를 이용하여 qPCR 정량분석을 2차에 걸쳐 수행하였다.
세균 군집 분포 특성 균주가 직접 토양으로 주입되는 관주 처리구에서 처리 후 6일과 13일 두 지점을 선정하여 유용균 처리에 따른 세균 군집 분포 특성의 변동을 평가하였다. 추출된 토양 DNA 염기서열은 Chunlab (Seoul, Korea)에서 Illumina Miseq. platform 장비를 이용하여 분석하였다. 시퀀싱은 16s rRNA V3-V4 region을 대상으로 수행되었으며, 프라이머와 바코드는 341F-805R을 사용하였다 (Fadrosh et al., 2014). 염기서열은 UPARSE 파이프라인을 이용하여 OTU 클러스터링 분석을 수행하였다. 염기서열들은 Mothur (version 1.27.0) 프로그램을 이용하여 분석하였으며 (Schloss and Handelsman, 2006), 기존에 표준화된 분석 파이프라인을 참조하여 염기서열을 정리하고 분석하였다 (Kim et al., 2012b; Choi et al., 2015). Good의 coverage, 종 풍부도 추정치, 다양성지수 및 벤다이어그램 (Venn diagram) 분석은 Mothur 프로그램을 이용하여 계산하였다 (Schloss et al., 2009). 유사성을 기반으로 한 시료간 베타 다양성 분석은 PCA 분석을 이용하였다.
토양 특성 평가 채취한 토양시료는 풍건하여 체 통과 (< 2 mm) 후 화학성 분석에 이용하였다. 토양 pH는 1:5 (토양:물) 비율로 침출하여 pH meter (Orion star A212, Thermo Scientific, Indonesia)로 측정하였다. 토양유기물은 Tyurin법을 이용하여 측정하였으며, 총질소함량은 켈달증류방법을 이용하여 분석하였다. 유효인산은 토양시료 5g에 Lancaster 용액 (pH 4.25) 20 mL를 넣은 후 10분간 진탕, 여과하여 UV/VIS Spectrometer (UV1900, Shimadzu, Kyoto, Japan)로 흡광도를 측정하여 분석하였다. 치환성 양이온은 1N NH4OAc로 침출하여 ICP-OES (Inductively coupled plasma-optical emission spectrophotometer, Perkin Elmer Model OPTIMA 4300DV, Norwalk, USA)를 이용하여 측정하였다 (NIAST, 1988).
통계 처리SAS package (SAS Institute Inc., 1995)를 이용하여 ANOVA 분석을 수행하였다. 균주처리에 따른 통계적인 유의성이 인정되는 경우, Tukey test를 이용하여 5% (p ≤ 0.05) 확률의 유의 수준에서 사후 분석을 수행하여 처리 간 효과를 비교하였다.
Results and Discussion
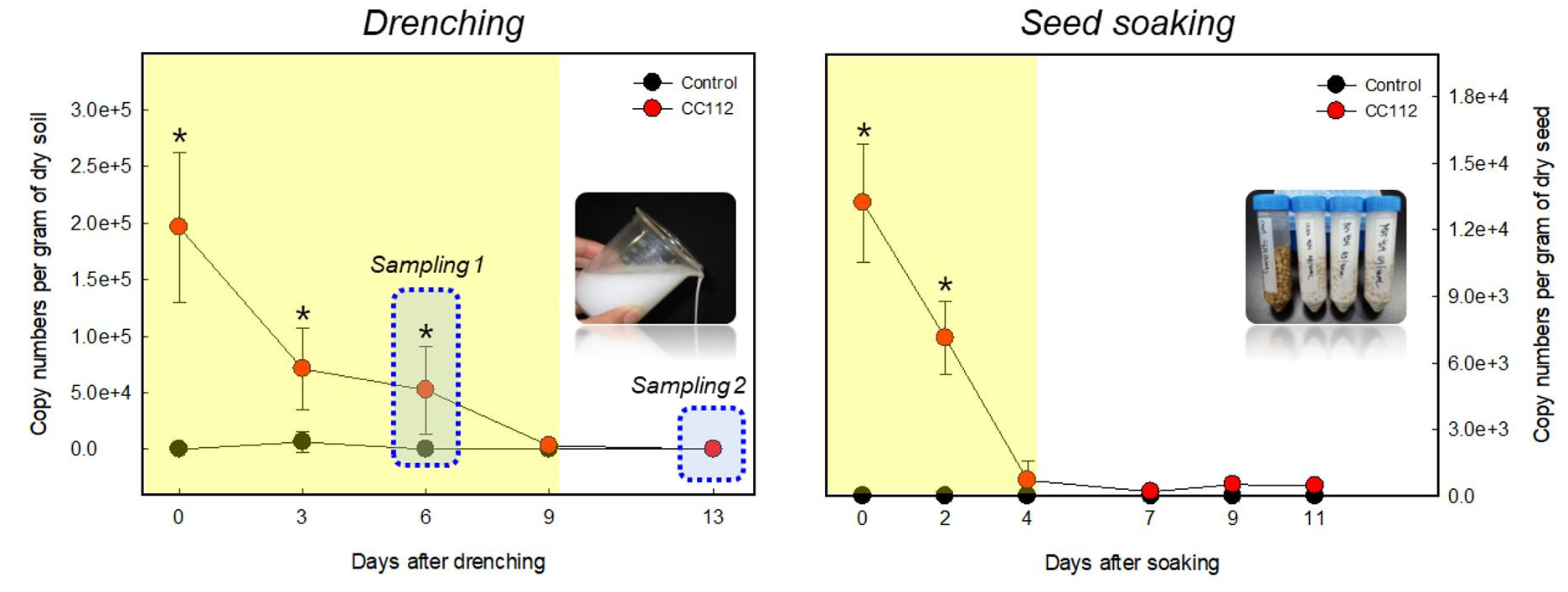
균주 처리방법별 토양 내 유용미생물 및 병원균 밀도 변화 균주 처리 방법에 따라 유용 균주 밀도는 약 4 - 9일 가량 토양에서 유지되는 것으로 나타났다 (Fig. 1). 관주 처리 직후 (2.03 × 105 copy numbers per gram of dry soil) 토양에서 약 9일 (3.2 × 103 copy numbers per gram of dry soil) 정도까지 유용균 밀도가 유지되는 것으로 분석되었다. 이전 연구에서 인삼 뿌리썩음병에 길항력이 있는 Bacillus amyloliquefaciens GR4-5 균주를 관주 처리 후 균주 밀도 변화를 조사한 결과, 실내 조건을 제외한 실외 및 포장시험에서는 Bacillus sp. group의 유전자 수는 균주 처리 후 약 7일 내외로 유용균 밀도를 유지하는 것으로 나타났다 (Kim et al., 2015). 인삼재배 토양 노지 조건에서 관주 처리를 하는 경우, 약 1주일 간격으로 지속적으로 처리를 해야 유용 균주 밀도를 유의한 수준으로 유지할 수 있을 것으로 판단한다. 종자 침지의 경우 적은 양이지만 처리 직후 (1.3 × 104 copy numbers per gram of dry seed) 균주 밀도가 높았으나, 점차 감소하였으며, 종자 침지 후 약 11일 이상 유지되는 것으로 나타났다. 하지만 4일 이후부터 무처리 (불검출) 대비 유의미한 차이를 보이지는 않았으나, 다소나마 유용균 밀도가 유지되는 것을 보아 매우 적은 밀도로 존재할 가능성이 높을 것으로 추정되나, qPCR 정량 한계 미만 (약 1,000 copy미만)으로 존재할 것으로 판단된다 (Skovhus et al., 2004). 향후 보다 정밀한 평가를 위해, FISH (fluorescent in situ hybridization) 등 다른 분자생물학적인 기법을 접목하여 유용 균주의 토양 및 종자 내 밀도와 정착능에 대한 추가 분석이 이루어져야 할 것으로 사료된다.
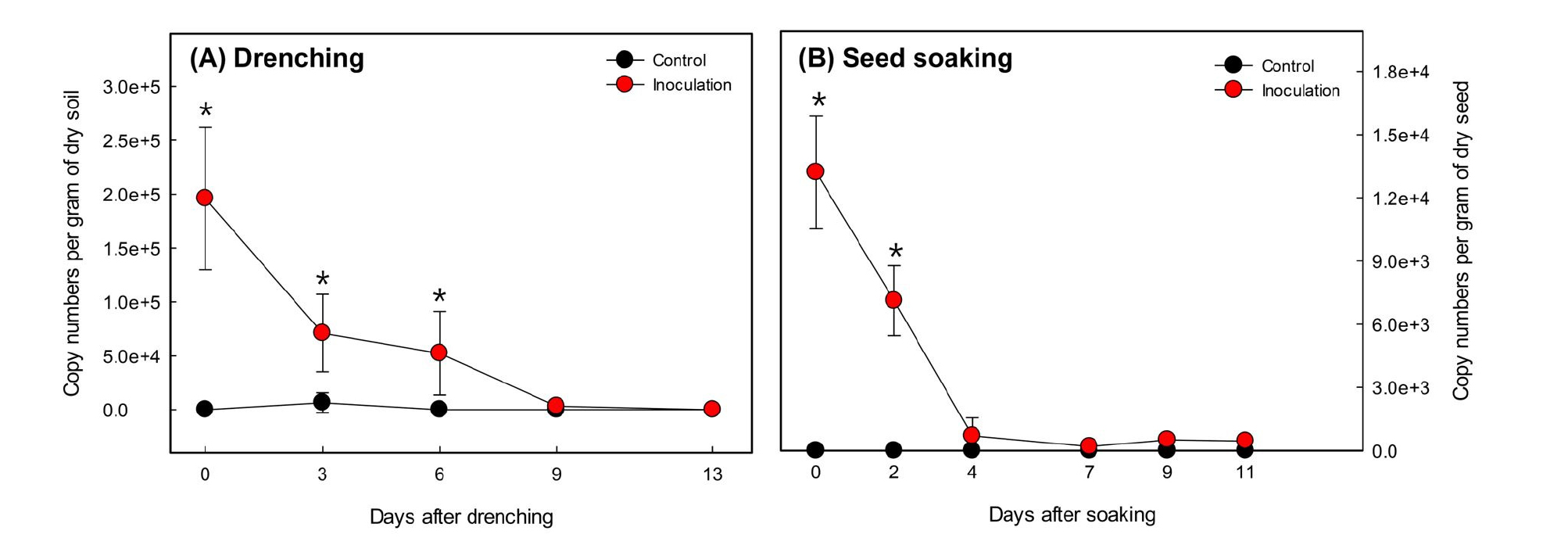
Fig. 1.
Changes in Bacillus abundance in ginseng soils after drenching CC112 (1/50 dilution of agent) as compared to the control treatment by water drenching (A) and seed soaking (B) methods in a greenhouse condition. The asterisks indicate significant differences between treatments at p ≤ 0.05 according to student’s t test. Bars represent standard deviation (n=3).
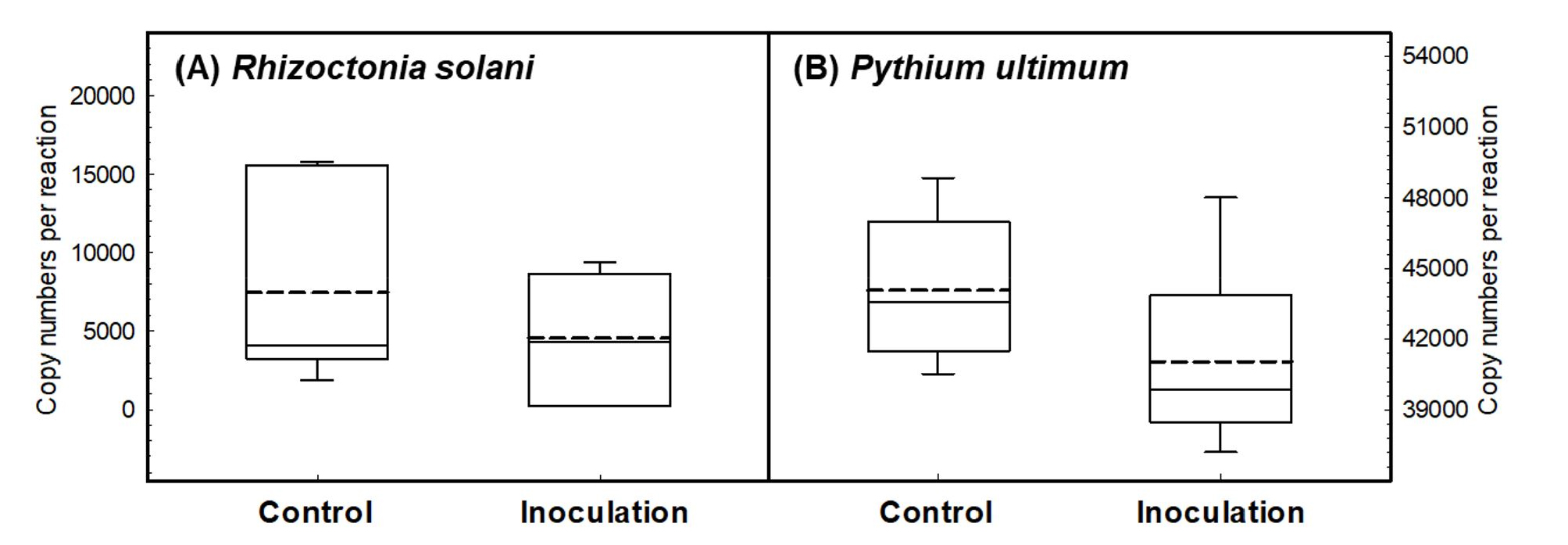
Nested qPCR을 활용하여 유용균 처리에 따른 토양 내 병원균의 밀도를 평가한 결과 무처리와 비교하여 Rhizoctonia solani는 약 39%, Pythium ultimum 의 경우 약 7% 수준에서 각각 인삼잘록병 유발 균주에 대한 방제 효과가 있는 것으로 나타났다 (Fig. 2). B. velezensis 의 경우 Fengycin 등과 같은 다양한 이차대사산물을 생산하여 병원성 곰팡이에 대한 항균활성을 지닐 수 있으며 (Kim et al., 2016; Kim et al., 2017aKim et al., 2017b), 본 연구에서 처리된 CC112 균주의 경우 분비되는 항균 물질의 생산으로 인해 식물병원성 곰팡이의 밀도가 다소 감소될 수 있었던 것으로 판단한다.
유용미생물 처리에 따른 토양 미생물 군집 분포 특성 유용균 처리에 따른 토양 미생물 종풍부도 (Ace, Chao)와 다양성 (Shannon, Invsimpson)은 관주 처리 초기 (6일)에 감소하는 양상이었으나, 13일 이후 무처리 토양과 동일한 수준으로 유의하게 회복되는 경향을 보였다 (Table 2). 본 결과를 바탕으로 유용미생물의 경우 관주 처리 후 초기에는 토양 미생물의 군집에 영향을 줄 수 있는 것으로 판단되나, 일정 시간이 지난 후 토양 세균 군집의 완충능력으로 인해 군집 구조가 원래 상태로 회복되는 것으로 해석된다 (Allison and Martiny, 2008).
Table 2.
Numbers of sequences, OTUs (97%), and diversity indices.
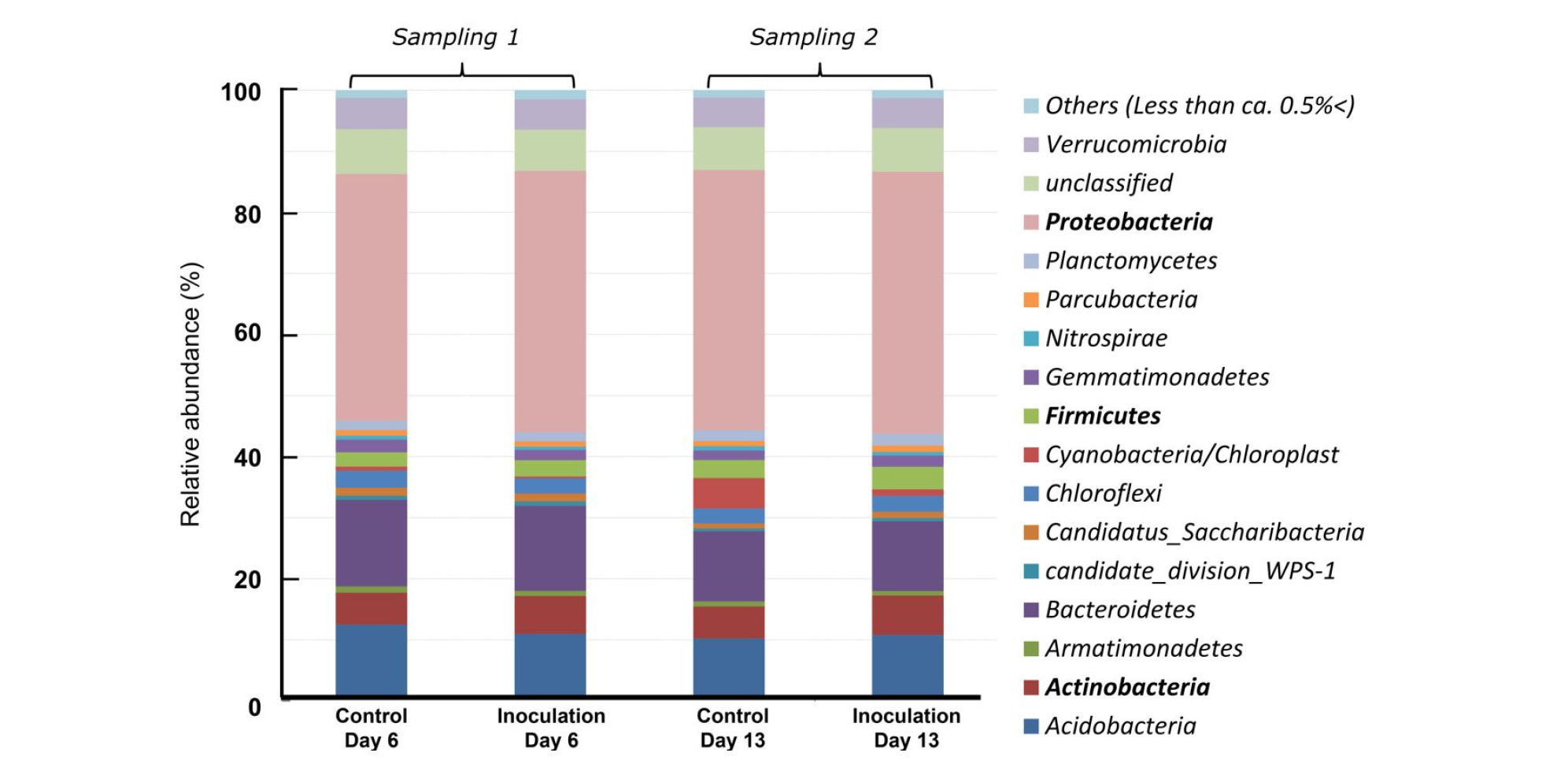
토양 전체 군집 분포를 자세히 조사하기 위해서 유전적으로 유사한 염기서열들을 종 (species) 단위 수준으로 묶어둔 분류 단위인 OTU (Operational taxonomic unit)를 주로 분석한다 (Edgar, 2013). 본 연구에서는 전체 조사 토양에서 2,366개 OTU를 공유하였고 (Fig. 3A), Proteobacteria가 주로 우점하는 OTU 인 것으로 나타났다. 균주 무처리 시료 (13일차)에서 나타나는 변화는 chloroplast의 오염으로 판단되어 우점 OTU 분포는 균주 처리에 따라 뚜렷한 변동을 보이지 않은 것으로 보인다 (Fig. 3B).
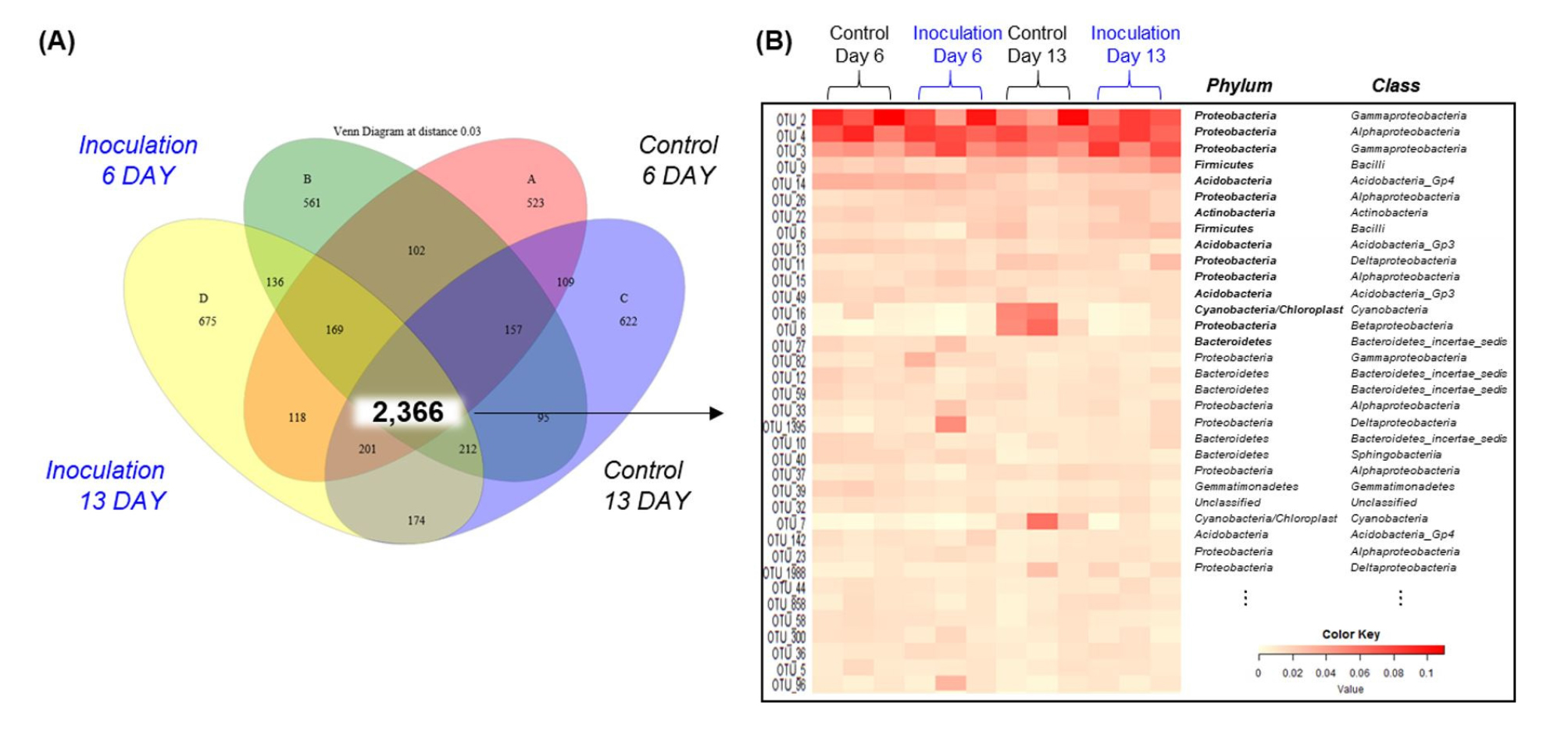
Fig. 3.
Core bacterial 2,366 OTUs merged in all samples. Venn diagram shows variable overlap between two sampling stages of ginseng soils after the inoculation of B. velezensis CC112 as compared to the control (A). The 2,366 operational taxonomic units (OTUs) were co-existed in all the stages. Heat map of the inoculation and sampling date displayed by core bacterial community (B).
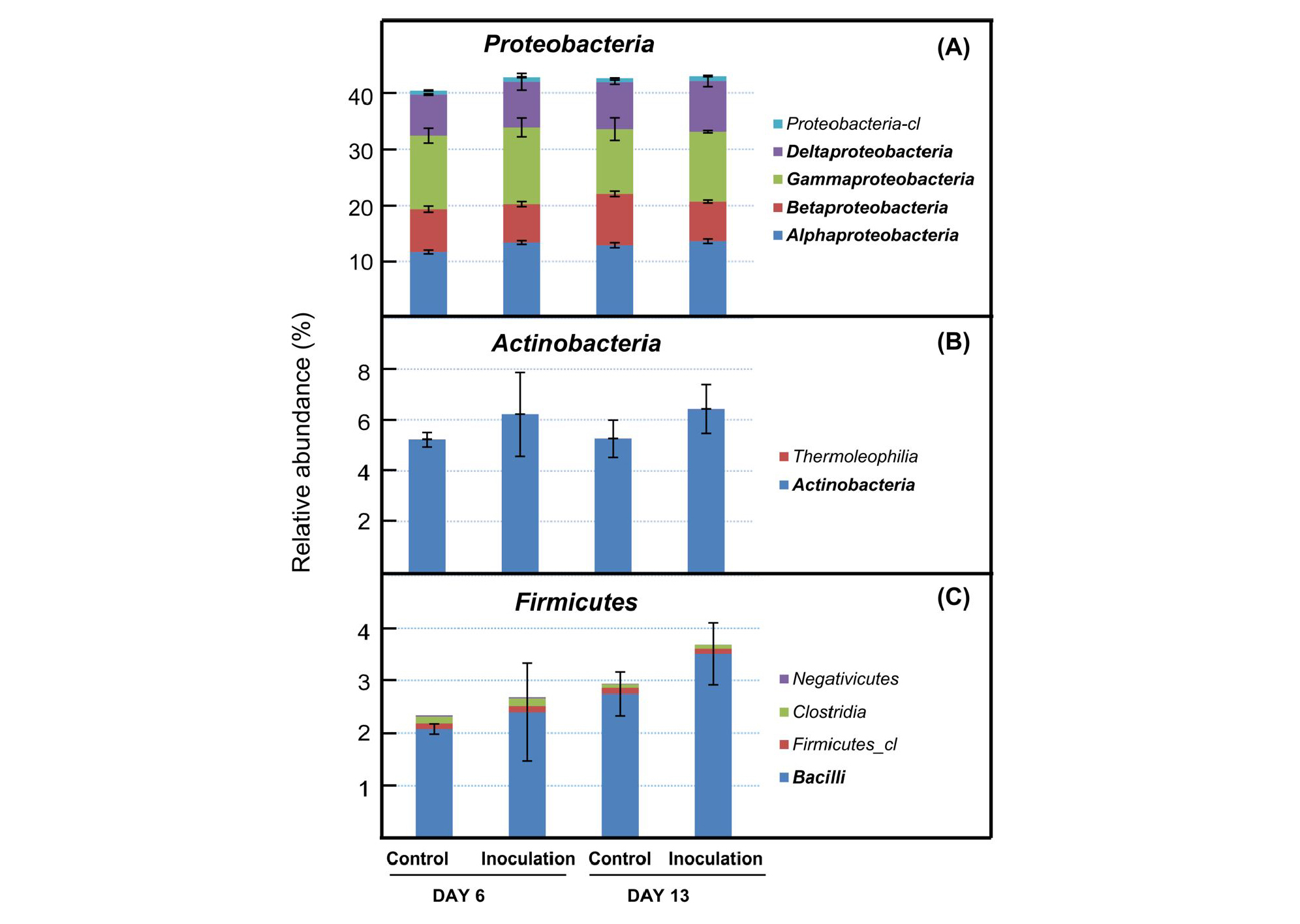
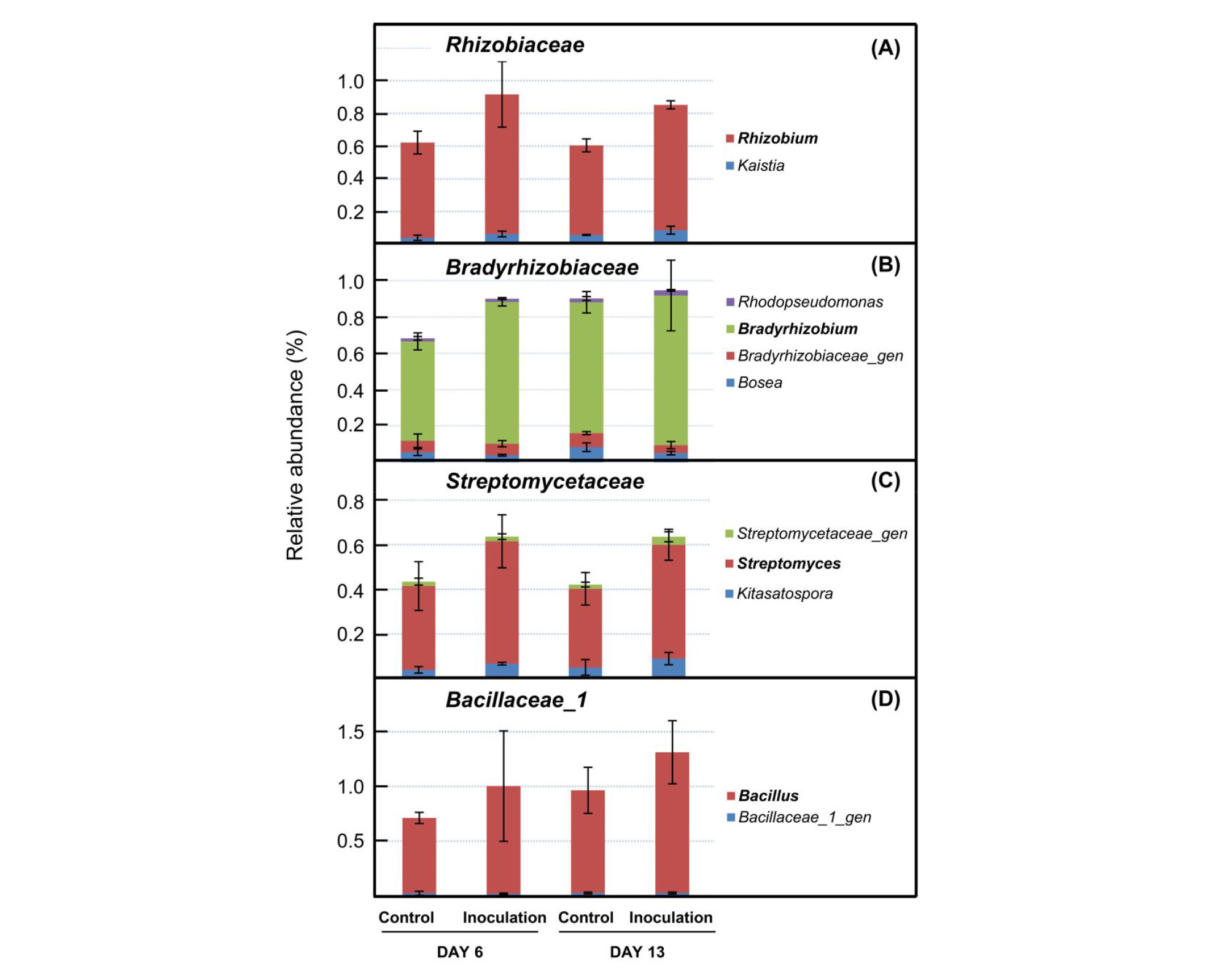
우점 군집은 phylum 수준에서는 Proteobacteria 인 것으로 조사되었으며, 일반적으로도 토양 내 phylum 수준에서 Proteobacteria 우점하는 것으로 알려져 있다 (Schloss and Handelsman, 2006). B. velezensis CC112 관주 처리 토양에서 주로 Proteobacteria, Firmicutes, Actinobacteria 의 비율이 증가하였다 (Fig. 4). 균주 처리에 따라 Proteobacteria 문에서는 Alphaproteobacteria, Deltaproteobacteria, Gammaprobacteria 강의 비율이 증가하는 경향을 보였지만, Betaproterobacteria 강의 비율은 다소 감소하였다 (Fig. 5). 선발균 처리에 따라 Actinobacteria 및 Bacilli 강의 비율 또한 증가하는 경향을 보였다. Genus 수준에서는 균주 처리에 따라 질소를 고정할 수 있는 것으로 알려져 있는 Rhizobium과 Bradyrhizobium의 비율이 상대적으로 증가되는 양상을 보였다. 일반적으로 Rhizobium과 Bradyrhizobium은 주로 콩과식물을 대상으로 공생적으로 질소를 고정하는 균으로서 연간 질소를 약 50 - 100 kg 가량 공급할 수 있다 (Graham and Vance, 2000). 질소를 고정할 수 있는 군집 비율의 증가가 토양에서 질소유효도를 증가시켜 인삼 생육 및 토양 환경에 긍정적인 영향을 줄 수 있을 것으로 판단된다 (Fig. 6). 그러나 인삼의 경우 콩과식물과 공생적으로 질소를 고정할 수 없는 점을 감안하면, 직접적인 토양 내 질소 공급 효과는 크지 않을 것으로 판단되나, 주변에 서식하는 콩과식물 잡초 등과 상호작용을 통해서 잠재적으로 인삼 생육에 긍정적인 영향을 줄 수 있을 것으로 판단한다. 항생물질 생산으로 인해 길항능력이 존재하는 것으로 보고된 Streptomyces (Ōmura et al., 2001) 및 Bacillus (Nakano and Zuber, 1990) 비율의 경우, 선발균 처리에 따라 증가되는 경향을 보였다. 궁극적으로 유용 균주 처리에 따라 토양 전체 미생물 군집 중에서 항생물질을 생산할 수 있는 군집의 비율이 상대적으로 증가한 것이 아마도 인삼잘록병 방제에 긍정적인 영향을 줄 수 있었던 것으로 판단한다.
Bray-curtis 분석 및 주성분 분석을 활용한 군집 특성 변화를 조사한 결과, 유용 균주 처리 후 초기 미생물 분포에서는 변동을 보였으나 이후 본래 토양의 특성으로 회복되는 경향을 보였다 (Fig. 7). 이는 토양이 지닌 완충능으로 인해 초기의 변화되었던 미생물 군집이 점차 원래의 특성으로 회복될 수 있는 것으로 판단된다 (Griffiths and Philippot, 2013). 본 결과를 바탕으로 유용균주의 처리는 원래 토양 미생물 군집 특성을 변화시키지 않으면서, 인삼잘록병을 효과적으로 방제할 수 있는 좋은 방제 기술이 될 것으로 본다.
유용미생물 처리에 토양환경 변동 평가 유용 미생물 관주처리에 따른 토양 화학성 변동을 평가하였다 (Table 3). 관주 처리구에서 토양의 특성이 무처리에 비해 전반적으로 개선되는 효과가 있는 것으로 보이나, 본 연구에서는 통계적인 유의차는 확인 할 수 없었다. 단, 관주처리구의 경우 유효 인산의 함량이 다소 높은 증가폭을 보였다. 토양 관주 처리를 통해 접종된 미생물과 다른 미생물 군집 간 양분 경쟁, 길항 작용 등 상호작용이 활발해지면서 상대적으로 Firmicute (Bacillus cereus, B. megaterium등)와 같은 미생물의 비율이 상대적으로 증가하면서 복합적으로 인산을 유효도를 높인 것으로 추정된다 (Lee et al., 2012). 본 연구결과를 통해 인산가용화능이 없거나 매우 낮은 단일 유용 균주 처리를 통해서도 유효 인산 함량 등을 포함한 토양 화학성을 개선할 수 있는 가능성이 다분히 존재하고 있음을 보여준다. 하지만 토양 내 인산 유효도 증가 및 토양 화학성 개선 효과에 대한 직접적인 원인에 대해서는 여전히 밝혀지지 않았다. 시료채취 혹은 분석 등의 오류를 감안하더라도 추후 관련 내용을 연구하여 밝혀낼 만한 충분한 가치가 있을 것으로 기대한다.
Conclusions
인삼 토양 내 유용균 관주 처리의 경우 약 1주일 간격으로 2 - 3회 이상 처리해야 균주 밀도를 일정 수준으로 유지할 수 있을 것으로 평가되었다. 유용균 처리에 따른 인삼잘록병 원인균의 밀도는 무처리 대비 Rhizoctonia solani는 약 39%, Pythium ultimum 의 경우 약 7% 수준에서 인삼잘록병 유발 균주에 대한 방제 효과가 있는 것으로 나타났다. 균주 처리에 따른 미생물 군집 특성 변동은 크지 않은 것으로 판단되며, Bacillus와 Streptomyces 속의 비율 증가가 아마도 인삼잘록병 방제에 긍정적인 영향을 줄 수 있었던 것으로 추정된다. 결론적으로 인삼 재배 토양 내 유용균 처리는 토양비옥도를 개선하고, 전체적인 토양미생물의 변동없이 인삼잘록병을 방제할 수 있는 유용한 방안이 될 수 있을 것으로 판단된다.